Computational Deconvolution for More Precise Biological Data
Deconvolution
Deconvolution 译作反卷积或解卷积。解卷积是一种基于算法的过程,用于反转卷积对记录数据的影响。
生物学数据,如 DNA expression、RNA sequencing、Protein expression 等,很难真正得到来自特定组织的“纯数据”。如血液中含有来自不同组织的细胞、外泌体等,由此得来的生物学数据将是不同类型细胞的混合数据。这时就需要通过解卷积,从混合的生物数据中解得不同类型细胞的比例、表达谱。
基本模型
测得的数据为不同类型细胞按照一定比例混合在一起的结果。
- 测得的表达矩阵为 X,则 xij 表示 genei 在 samplej 中的表达量或表达强度
- 比例矩阵为 R,则 rkj 表示 k 类细胞在 samplej 中所占比例
- 各细胞真实表达谱组成的矩阵为 S,则 sik 表示表示 genei 在 k 类细胞中的表达量或表达强度

解卷积算法
针对不同情况有不同的算法,根据项目中的先验知识选择合适的解卷积方法以达到最佳效果。
生物数据解卷积分为完全解卷积和不完全解卷积。
- 完全解卷积:指混合细胞类型数目、比例和各细胞表达谱未知,仅依赖混合表达数据将它们解出来,即已知混合表达矩阵 X,解比例矩阵 R 和各细胞表达矩阵 S。完全解卷积的完全指细胞表达谱和比例都在算法中一次性解得。
- 不完全解卷积:指已知混合表达谱、细胞类型数目、比例,解各细胞表达谱,即已知比例矩阵 R 和混合表达矩阵 X,解矩阵各细胞表达矩阵 S。不完全是相对于完全解卷积而言,仅解得各细胞表达谱。
完全解卷积
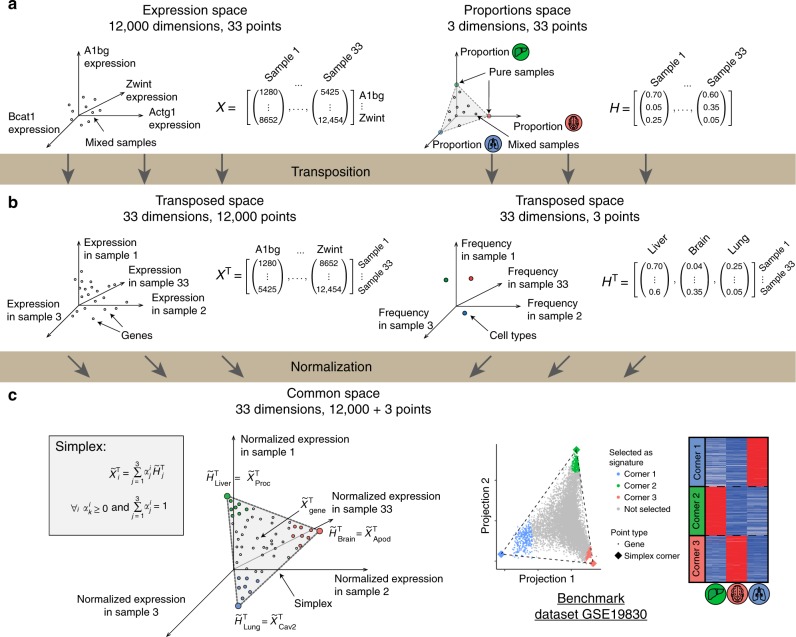
LinSeed
通过计算共线性网络 (Collinearity Network) 得到各类型细胞的 markers,利用 markers 对混合数据进行完全解卷积,同时得到各组分的比例和表达谱。
Code availability:
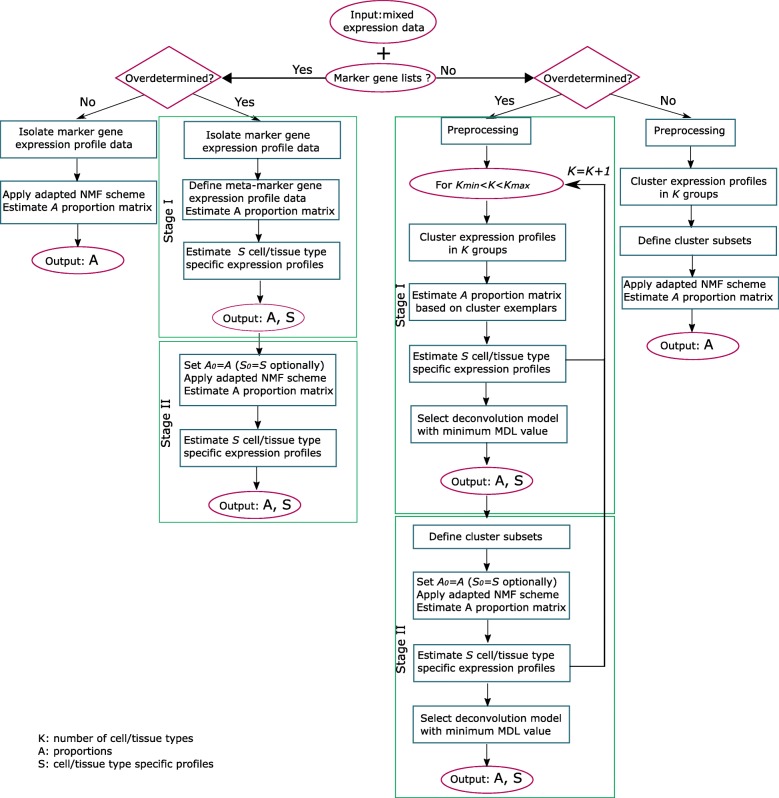
Deblender
Deblender 是一个灵活的完全去卷积工具。基于用户对已知标记基因列表和细胞/组织组成信息的访问 (access),以半/无监督模式运行。在没有先验知识的情况下,全局基因表达的变异性被用于混合数据的聚类,以聚类集代替标记集。
Code availability:
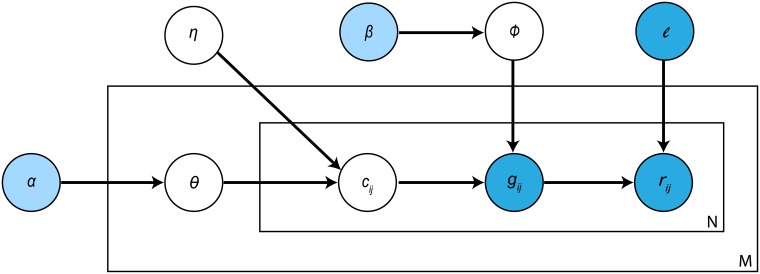
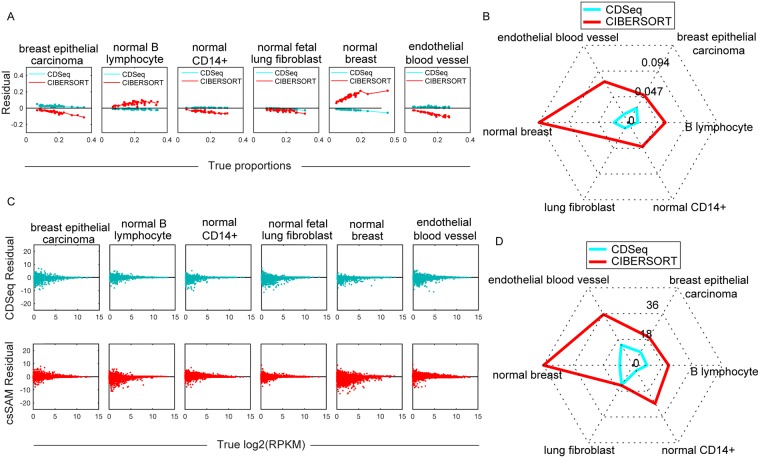
CDSeq
CDSeq 仅使用来自大量组织样本的 RNA-Seq 数据来同时估计细胞类型比例和细胞类型特异性表达谱。
Code availability:
- MATLAB and Octave
- https://github.com/kkang7/CDSeq
Deconf
Deconf 证明了从单个样本的基因表达数据中预测构成细胞类型的比例的可行性,这是基于去粗取精的分类方法的前提条件。
Code availability:
不完全解卷积
Rodeo
Rodeo 是一种基于稳健线性回归的方法,可以实现简单而稳健的表达式反卷积。
Code availability:
cs-lsfit & cs-qprog
cs-lsfit 和 cs-qprog 属于 CellMix 中的两个解卷积算法,使用CellMix时可选择使用哪一个来进行解卷积步骤。
Code availability:
LRCDE
LRCDE 在逐个基因的基础上执行基于线性回归的细胞类型特异性差异表达(反卷积)检测。考虑到细胞类型特异性基因表达估计的变异性,它计算差异检测的每个基因 t 统计量、p 值、基于 t 统计量的灵敏度、组特异性均方误差和几个基因特异性诊断指标。
Code availability:
csSAM
csSAM 根据测量的细胞比例计算细胞特异性差异表达。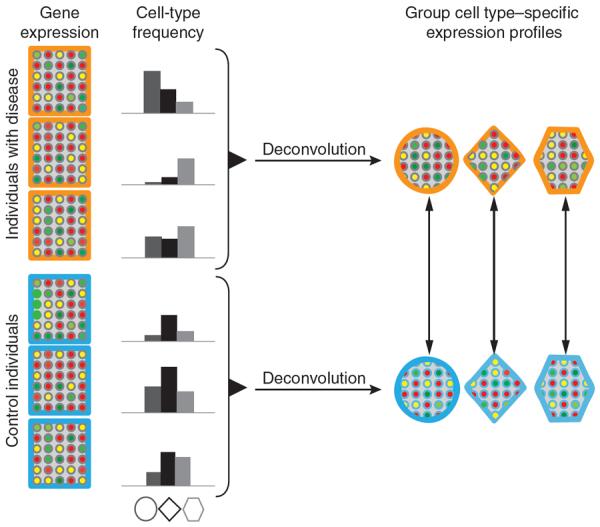
Code availability:
参考文献
[1] Maria K Jaakkola, Laura L Elo, Computational deconvolution to estimate cell type-specific gene expression from bulk data, NAR Genomics and Bioinformatics, Volume 3, Issue 1, March 2021, lqaa110, https://doi.org/10.1093/nargab/lqaa110
[2] 具体算法参考文献见算法介绍部分